Polymer design via SHAP and Bayesian machine learning optimizes pDNA and CRISPR ribonucleoprotein delivery
논문 정보
- 제목: Polymer design via SHAP and Bayesian machine learning optimizes pDNA and CRISPR ribonucleoprotein delivery
- 저자: Rishad J. Dalal (University of Minnesota), Felipe Oviedo (Nanite Inc., University of Minnesota), Michael C. Leyden (University of Minnesota), Theresa M. Reineke (University of Minnesota)
- 학회/저널: Chemical Science
- 발행일: 2024-04-22
- DOI: 10.1039/d3sc06920f
- 주요 연구 내용: 길이, 이원 조성, , 소수성(clog P)을 체계적으로 변화시킨 폴리머 라이브러리를 합성함. SHAP(SHapley Additive exPlanations) 분석과 베이지안 최적화(BO) 머신러닝을 결합하여 plasmid DNA(pDNA) 및 CRISPR-Cas9 리보핵단백질(RNP) 전달을 위한 정량적 구조-특성 관계를 해석하고 최적의 제형을 예측함.
- 주요 결과 및 결론: pDNA 전달에는 낮은 폴리머 와 높은 benzimidazole ethanethiol(BET) 비율이 유리한 반면, RNP 전달에는 더 긴 폴리머 길이와 captamine(Cap) 양이온이 더 효과적임을 발견함. 머신러닝으로 최적화된 상위 3개 폴리머는 생체 내(in vivo)에서 대조군보다 1.7배 향상된 유전자 발현을 보이며 20일 이상 안정적으로 유지됨.
- 기여점: 핵산의 종류에 따라 최적의 전달체 설계 파라미터가 다르다는 것을 설명 가능한 머신러닝(SHAP)을 통해 처음으로 규명함. 합성, 특성 분석, 머신러닝을 결합한 파이프라인을 제시하여 차세대 핵산 약물 전달체 개발을 가속화할 수 있는 강력한 도구를 제공함.
요약
초록
길이, 조성, , 소수성이 다른 폴리머 라이브러리를 합성하여 pDNA와 CRISPR-Cas9 RNP의 세포 내 전달 성능을 최적화했다. 머신러닝을 통해 432개 제형을 분석한 결과, pDNA 전달에는 낮은 폴리머 와 높은 benzimidazole ethanethiol(BET) 비율이, RNP 전달에는 긴 폴리머 길이와 captamine 양이온이 중요하다는 상반된 설계 파라미터를 발견했다. 베이지안 최적화를 통해 시험관 내 성능을 더욱 향상시켰고, 최종 선별된 상위 3개 폴리머는 생체 내 실험에서 대조군보다 1.7배 향상된 성능과 20일 이상의 안정적인 유전자 발현을 보였다. 이 연구는 합성, 특성 분석, 머신러닝을 결합하여 차세대 핵산 약물 전달체 개발을 가속화하는 방법을 제시한다.
서론
핵산 치료제는 유망한 치료법이지만, 전달 효율 문제로 임상 적용에 어려움을 겪고 있다. 바이러스성 전달체의 한계를 극복하기 위한 대안으로 비바이러스성 전달체인 고분자가 주목받고 있다. 고분자는 화학적/물리적 변형이 쉽고 저렴하게 대량 생산이 가능해 잠재력이 크지만, 아직 성능이 낮아 활용이 저조하다. 이 연구에서는 병렬 실험과 머신러닝을 결합하여 방대한 화학 공간에서 최적의 고분자 전달체를 효율적으로 탐색하고, pDNA와 CRISPR RNP 전달에 영향을 미치는 핵심 물리화학적 특성을 규명하고자 한다.
모델 아키텍처 / 방법론
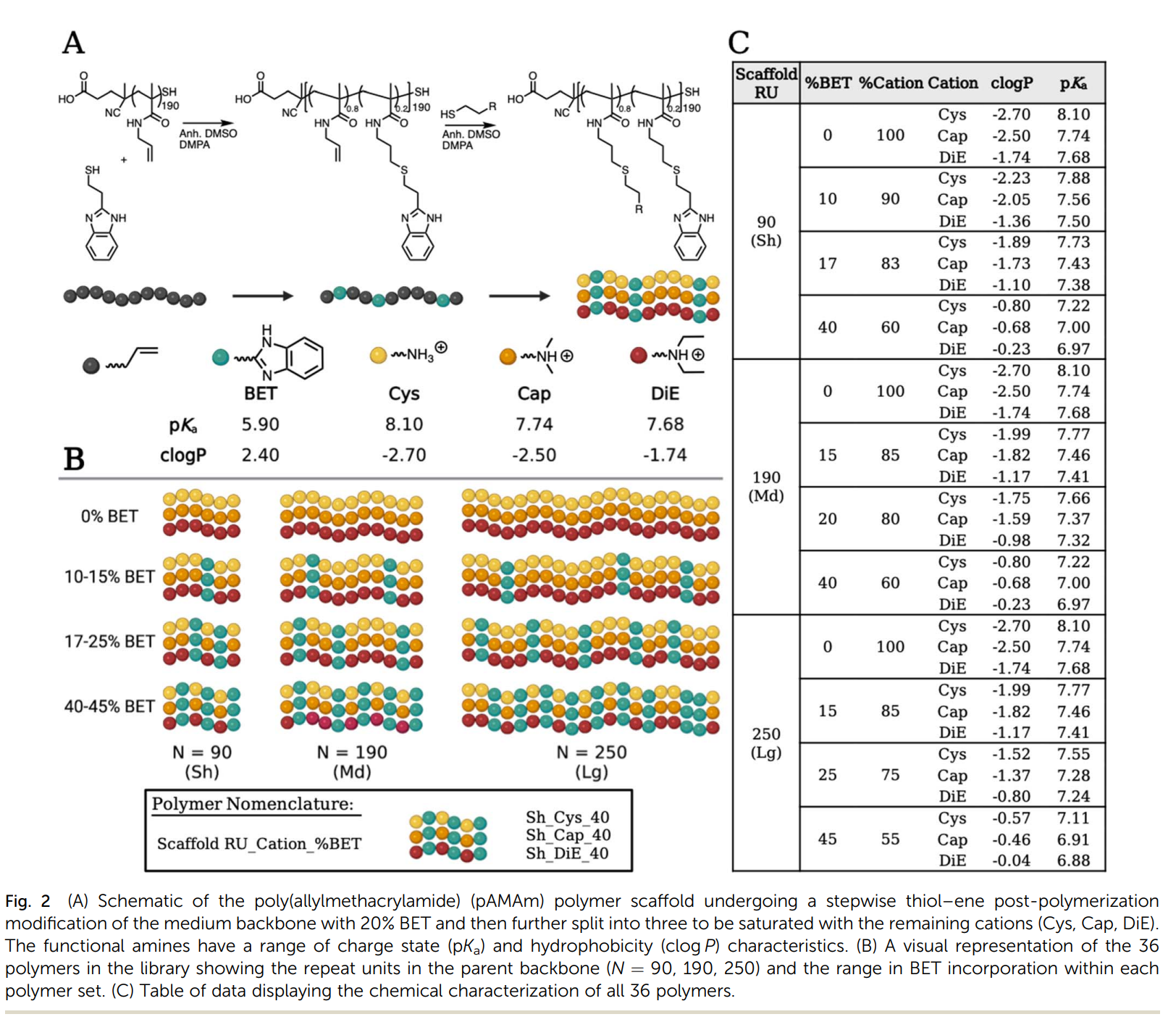
- 핵심 구조/방법: RAFT 중합을 통해 세 가지 길이(Sh, Md, Lg)의 고분자 골격을 합성한 후, Thiol-ene 클릭 화학을 이용해 소수성 양이온인 BET와 다른 세 종류의 공양이온(Cys, Cap, DiE)을 체계적인 비율로 도입했다. 이를 통해 총 36개의 공중합체 라이브러리를 구축하여 물리화학적 특성을 조절했다. 이 과정은 논문의 Figure 2A에 도식화되어 있다.
- 주요 구성 요소:
- 고분자 골격 (Scaffold RU): 짧은(Sh, N=90), 중간(Md, N=190), 긴(Lg, N=250) 세 종류의 길이를 가진다.
- 양이온 (Cations): 소수성을 부여하는 Benzimidazole ethanethiol (BET)과, 정전기적 결합을 촉진하는 Cysteamine (Cys), Captamine (Cap), 2-(diethylamino)ethanethiol (DiE)을 사용했다.
- 물리화학적 특성: 각 고분자의 양성자화 상태()와 소수성(clog P)을 측정하고 이를 핵심 변수로 활용했다.
- 알고리즘:
- SHAP (SHapley Additive exPlanations): 고분자의 특성(길이, 양이온 종류, BET 비율, 등)이 유전자 발현 및 세포 생존율에 미치는 영향을 정량적으로 분석하여 각 특성의 중요도를 파악했다.
- 베이지안 최적화 (Bayesian Optimization, BO): SHAP 분석 결과를 바탕으로, 제한된 수의 실험만으로 최적의 제형 비율(N/P ratio)을 효율적으로 예측하고 검증했다.
실험 결과
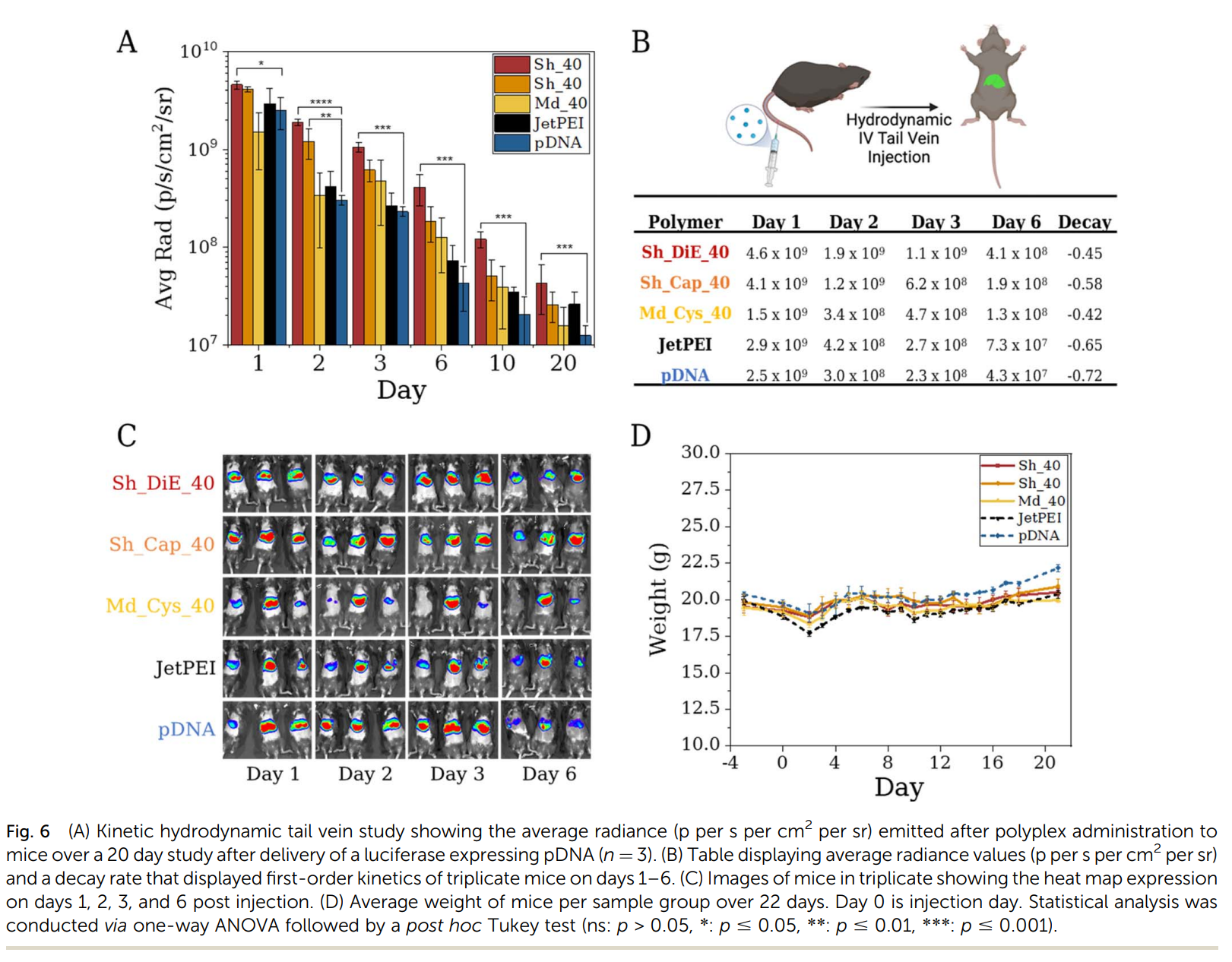
- 주요 데이터셋: HEK293T 세포를 사용하여 pDNA(GFP 발현) 및 RNP(mCherry 발현) 전달 효율과 세포 독성을 평가했다. 최종 선별된 고분자는 마우스 모델에 정맥 주사하여 생체 내 루시퍼라아제 발현 효능을 장기간 관찰했다.
- 핵심 성능 지표:
- pDNA 전달: Sh*DiE_40 폴리머가 N/P=10에서 75%의 GFP 발현율을 보여 대조군(JetPEI, 77%)과 유사한 최고 성능을 보였다. SHAP 분석 결과, 낮은 와 높은 BET 비율이 GFP 발현에 긍정적인 영향을 미쳤다.
- RNP 전달: Lg_Cap_0 폴리머가 N/P=5에서 약 6%의 mCherry 발현율을 보여 대조군(JetPEI, 1%)보다 6배 높은 성능을 보였다. SHAP 분석 결과, 긴 폴리머 길이와 Cap 양이온이 RNP 전달에 더 중요했다.
- 생체 내(In vivo) 결과: SHAP과 BO로 선별된 상위 폴리머들은 20일 동안 대조군보다 높고 안정적인 유전자 발현을 유지했다. 특히, 발현 감소율이 대조군보다 1.7배 낮아 장기적인 효과를 보였다. 실험 결과 그래프는 논문의 Figure 6A에서 확인할 수 있다.
- 비교 결과: pDNA와 RNP 전달에 유리한 고분자의 화학적 특성이 명확히 달랐다. pDNA 전달에는 소수성(BET)과 낮은 가 중요했지만, RNP 전달에는 긴 사슬 길이와 특정 양이온(Cap)이 더 중요했으며, BET는 오히려 성능을 저해하는 경향을 보였다.
결론
체계적인 고분자 합성, 병렬 스크리닝, 그리고 머신러닝(SHAP, BO)을 결합한 효율적인 워크플로우를 성공적으로 구축했다. 이 접근법을 통해 pDNA와 RNP라는 서로 다른 핵산 전달에 최적화된 고분자 설계 원리가 다르다는 것을 정량적으로 규명했다. 본 연구에서 제시된 방법론은 방대한 후보 물질 중에서 최적의 전달체를 효율적으로 발굴하여 차세대 핵산 치료제 개발을 가속화할 수 있는 강력한 플랫폼을 제공한다