Transfer Learning with Self-Supervised Vision Transformer for Large-Scale Plant Identification
논문 정보
- 제목: Transfer Learning with Self-Supervised Vision Transformer for Large-Scale Plant Identification
- 저자: Mingle Xu (Jeonbuk National University), Sook Yoon (Mokpo National University), Yongchae Jeong (Jeonbuk National University), Jaesu Lee (Rural Development Administration), Dong Sun Park (Jeonbuk National University)
- 학회/저널: CLEF 2022
- 발행일: 2022-09-05
- DOI: N/A
- 주요 연구 내용: 대규모 소수샷(few-shot) 식물 식별 챌린지인 PlantCLEF2022를 해결하기 위해, 기존의 지도 학습 방식 CNN 대신 자기 지도 학습으로 사전 학습된 Vision Transformer(ViT) 모델, 구체적으로 Masked Autoencoder (MAE)를 사용함. 이 접근법은 ViT의 높은 모델 용량과 자기 지도 학습의 범용적인 특징 추출 능력을 활용함.
- 주요 결과 및 결론: 제안된 방법으로 PlantCLEF2022 챌린지에서 MA-MRR 0.62692를 기록하며 1위를 차지함. 추가 학습을 통해 0.64079까지 성능을 향상시켰으며, PlantCLEF2022 데이터셋으로 사전 학습된 모델이 다른 식물 질병 인식 과제에서도 성능 향상에 기여함을 보임.
- 기여점: 자기 지도 학습 ViT가 대규모, 소수샷 이미지 분류 문제에 효과적임을 입증함. 또한, PlantCLEF2022 데이터셋과 이를 통해 사전 학습된 모델이 식물 관련 하위 과제(downstream task)에 유용한 공개 자원이 될 수 있음을 시사함.
요약
초록
PlantCLEF2022 챌린지는 수백만 개의 이미지와 8만 개의 클래스를 가진 대규모 식물 식별 과제이지만, 클래스당 평균 이미지가 36개에 불과한 소수샷 학습 문제이다. 이 문제를 해결하기 위해, 일반적인 지도 학습 기반 CNN 대신 자기 지도 학습으로 사전 학습된 Vision Transformer(ViT)를 활용했다. 이 접근법을 통해 2위보다 0.019, 3위보다 0.116 높은 MA-MRR 0.62692로 1위를 달성했다. 논문은 자기 지도 학습 ViT의 우수성과 PlantCLEF2022 데이터셋의 활용 가능성을 강조하며, 사전 학습된 모델이 식물 질병 인식과 같은 관련 분야에도 기여할 수 있음을 보여준다.
서론
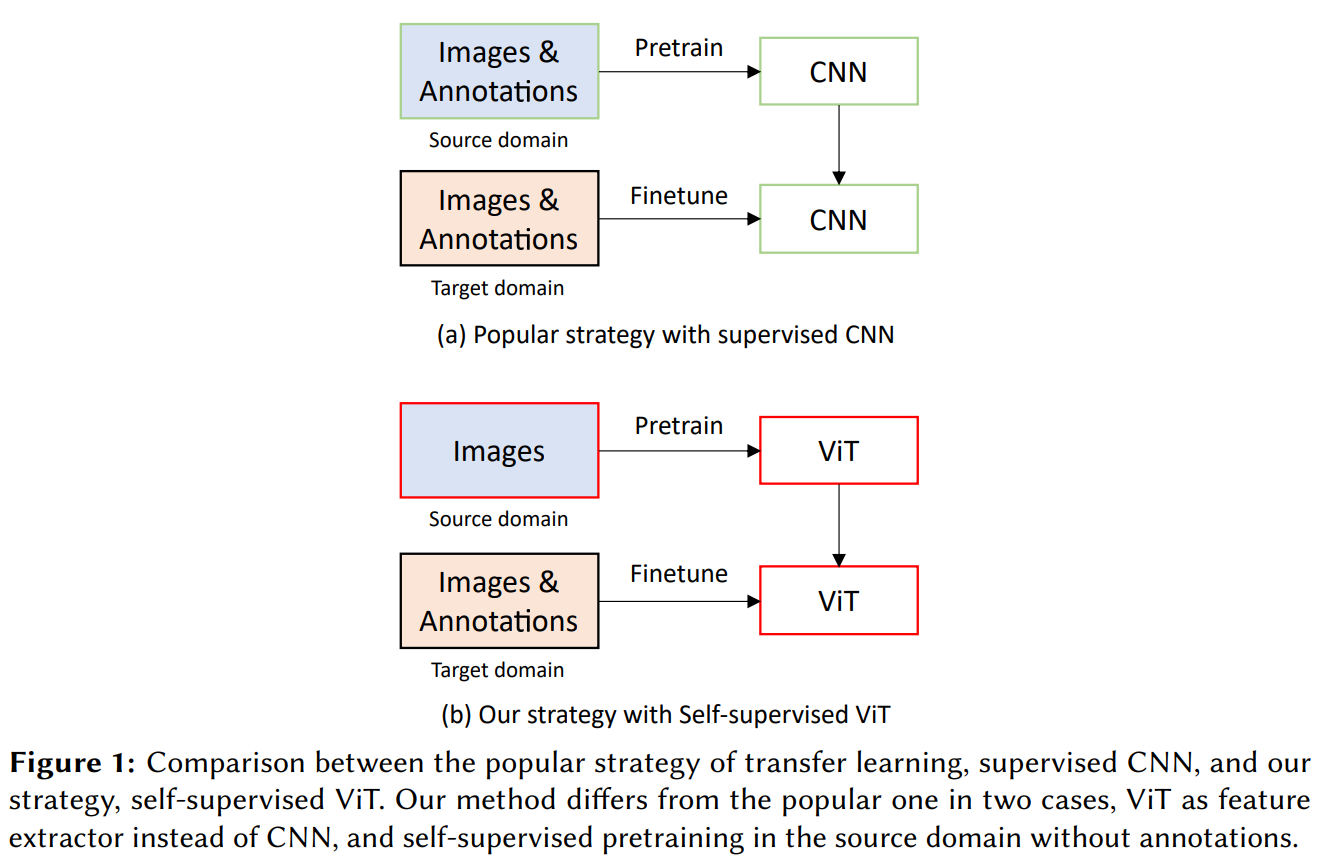 식물 종 식별은 생물 다양성 유지에 중요하지만 전문가의 많은 시간과 비용을 요구한다. 딥러닝 기반 자동 분류가 대안으로 떠올랐고, PlantCLEF2022는 이를 위한 대규모 챌린지이다. 일반적인 전이 학습 전략인 'ImageNet에서 지도 학습된 CNN'은 두 가지 약점을 가진다. 첫째, ImageNet과 식물 데이터셋 간의 도메인 차이로 인해 특징이 잘 전이되지 않을 수 있다. 둘째, CNN의 귀납적 편향(inductive bias)으로 인해 모델 용량이 제한된다. 이 연구는 이러한 약점을 극복하기 위해 자기 지도 학습(SSL)과 Vision Transformer(ViT)를 결합한 전략을 제안한다. 논문의 Figure 1은 기존 전략과 제안 전략을 시각적으로 비교한다.
식물 종 식별은 생물 다양성 유지에 중요하지만 전문가의 많은 시간과 비용을 요구한다. 딥러닝 기반 자동 분류가 대안으로 떠올랐고, PlantCLEF2022는 이를 위한 대규모 챌린지이다. 일반적인 전이 학습 전략인 'ImageNet에서 지도 학습된 CNN'은 두 가지 약점을 가진다. 첫째, ImageNet과 식물 데이터셋 간의 도메인 차이로 인해 특징이 잘 전이되지 않을 수 있다. 둘째, CNN의 귀납적 편향(inductive bias)으로 인해 모델 용량이 제한된다. 이 연구는 이러한 약점을 극복하기 위해 자기 지도 학습(SSL)과 Vision Transformer(ViT)를 결합한 전략을 제안한다. 논문의 Figure 1은 기존 전략과 제안 전략을 시각적으로 비교한다.
모델 아키텍처 / 방법론
-
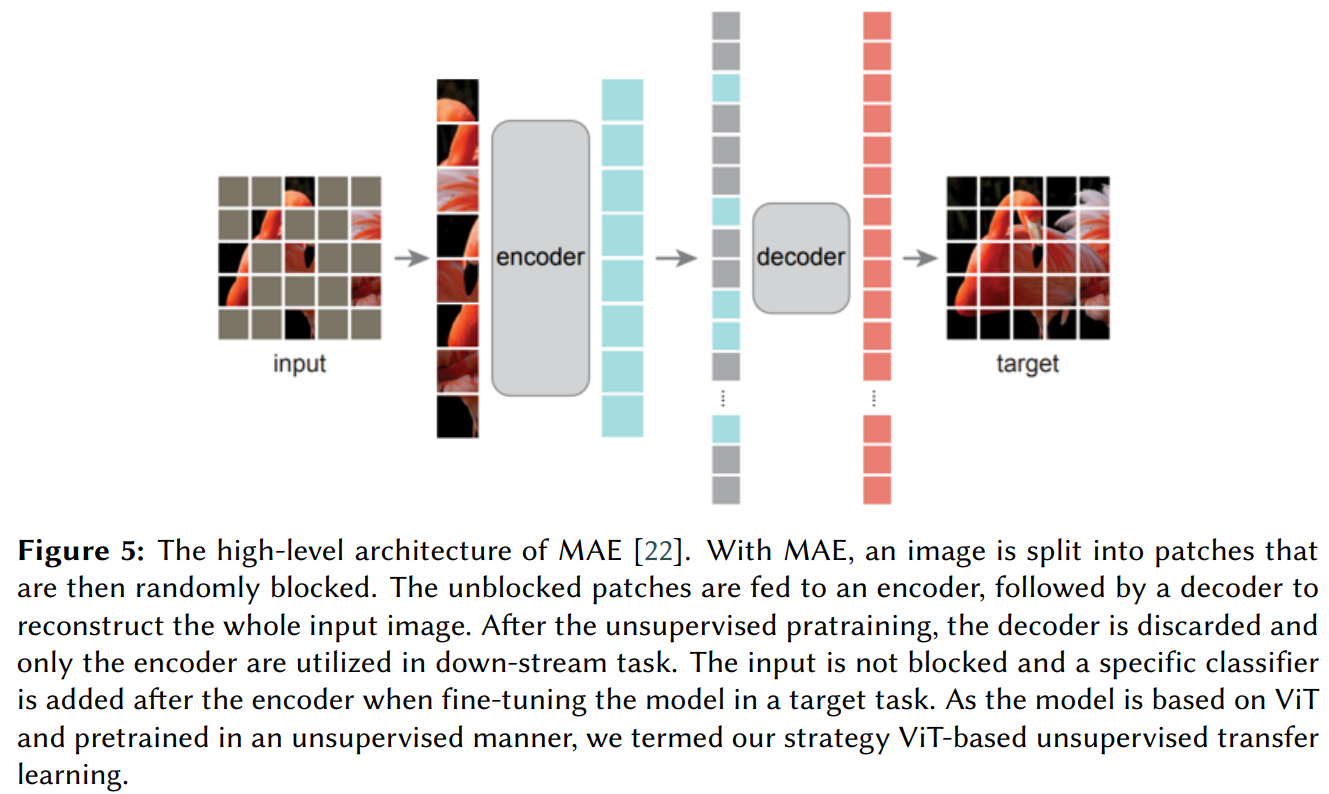
핵심 구조/방법: ImageNet1k에서 자기 지도 학습 방식으로 사전 학습된 Masked Autoencoder(MAE) 모델(ViT-Large 아키텍처 기반)을 PlantCLEF2022 데이터셋에 파인튜닝하는 전이 학습 전략을 사용했다. MAE는 이미지 패치를 무작위로 마스킹하고, 보이는 패치만 인코더에 입력하여 전체 이미지를 복원하도록 학습한다. 논문의 Figure 5는 MAE의 아키텍처를 보여준다.
-
주요 구성 요소:
- Encoder: ViT-Large 아키텍처. 마스킹되지 않은 패치로부터 특징을 추출한다.
- Decoder: 사전 학습 시에만 사용되며, 인코더의 특징을 바탕으로 마스킹된 패치를 포함한 전체 이미지를 복원한다. 파인튜닝 시에는 제거된다.
- Classifier Head: 파인튜닝 시 인코더 뒤에 추가되는 선형 레이어로, 80,000개의 클래스를 분류한다.
-
수식: 성능 평가 지표로 MA-MRR(Macro Averaged Mean Reciprocal Rank)을 사용했다. 여기서 은 클래스 수, 은 클래스 의 관찰(observation) 수, 는 번째 관찰에 대한 첫 번째 정답 레이블의 순위를 의미한다.
-
알고리즘: 관찰(Observation) 단위 분류를 위해 여러 이미지의 예측 결과를 통합해야 한다. 'Single-highest'(가장 높은 top-1 점수를 가진 이미지의 예측 사용)와 'Multi-sorted'(모든 이미지의 예측 점수를 정렬하여 상위 랭크 생성)와 같은 통합 전략을 사용했다.
실험 결과
-
주요 데이터셋: PlantCLEF2022의 'trusted' 데이터셋(이미지 290만 개, 클래스 8만 개)을 학습에 사용했다. 테스트셋은 26,868개의 관찰에 속한 55,306개의 이미지로 구성된다.
-
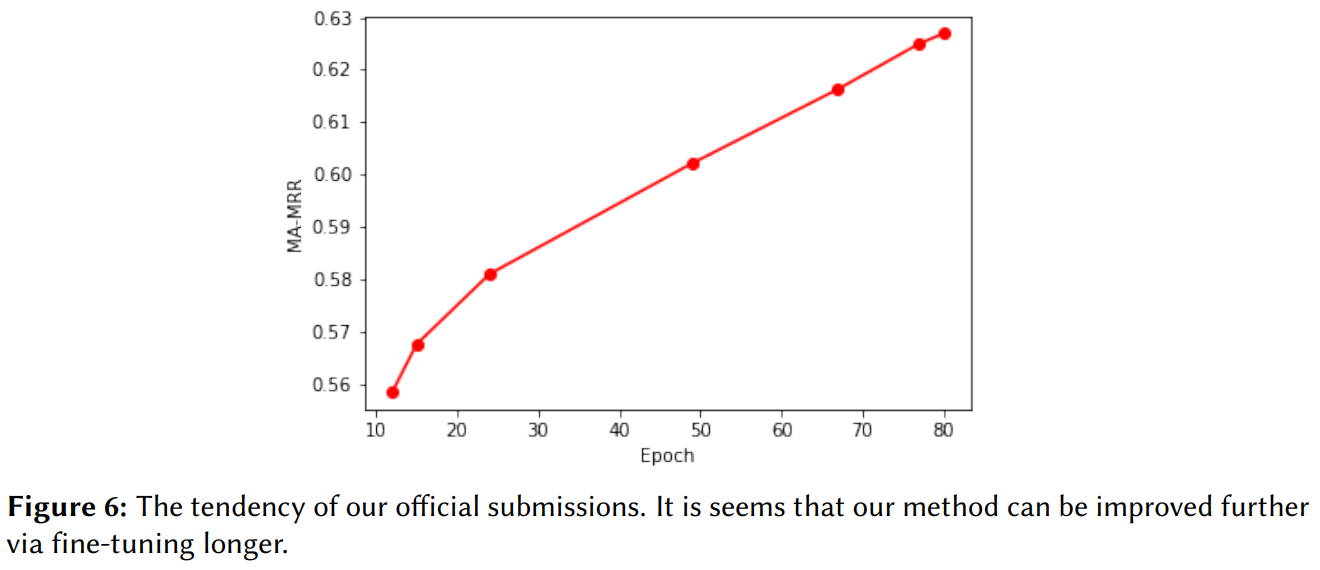
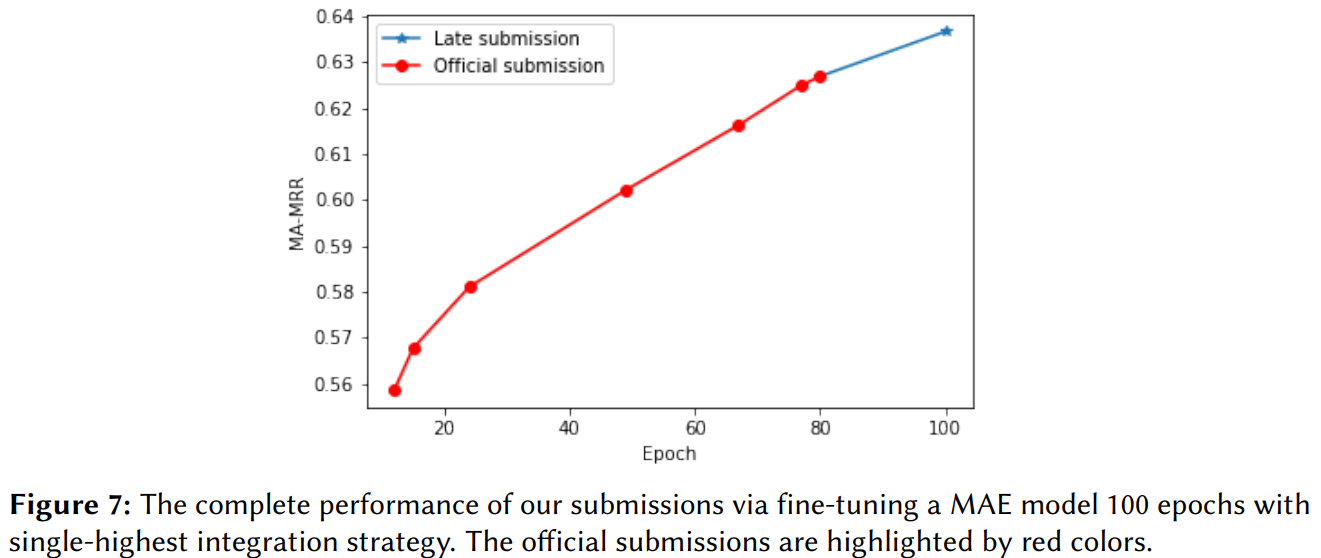
핵심 성능 지표: 80 에폭 학습 후 MA-MRR 0.62692를 달성하여 챌린지에서 1위를 차지했다. 학습을 100 에폭까지 연장했을 때 'Multi-sorted' 통합 전략으로 MA-MRR 0.64079를 기록했다. 논문의 Figure 6과 7은 에폭 증가에 따른 성능 향상 추세를 보여준다.
-
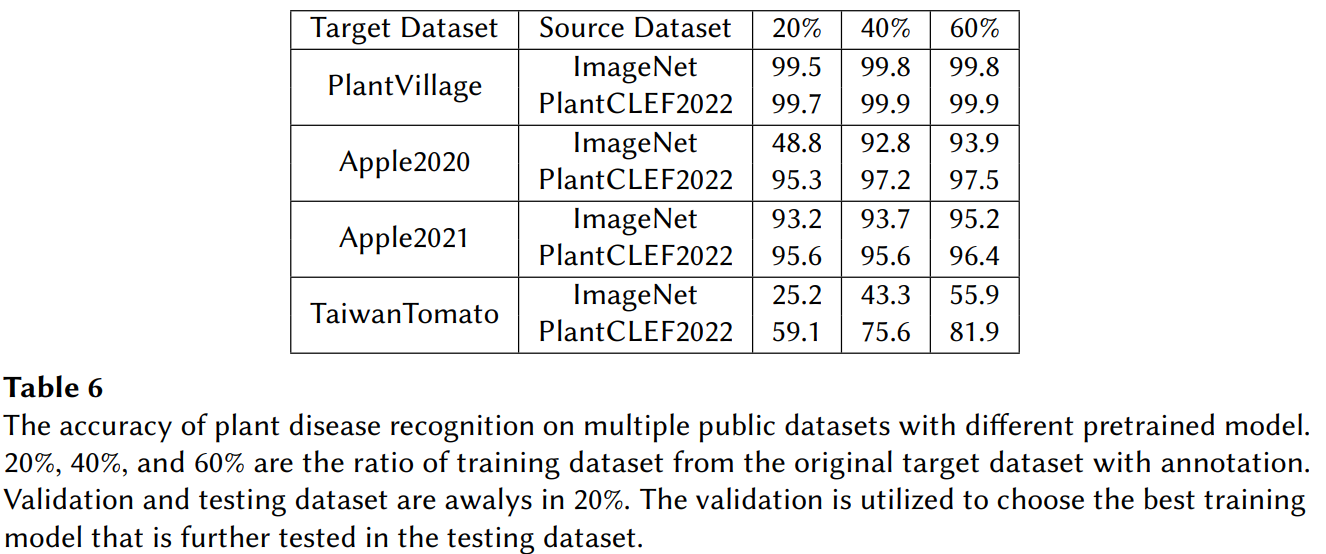
비교 결과: 공식 제출 결과(MA-MRR 0.62692)는 2위 팀(0.60781)과 3위 팀(0.51043)을 큰 차이로 앞섰다. 또한 PlantCLEF2022로 사전 학습한 모델을 4개의 공개 식물 질병 데이터셋에 적용한 결과, ImageNet으로만 사전 학습한 모델보다 더 빠르고 높은 정확도를 보였다 (Table 6 참조).
결론
이 연구는 자기 지도 학습으로 사전 학습된 Vision Transformer가 대규모 소수샷 식물 식별 과제에 매우 효과적인 접근법임을 입증했다. 기존의 지도 학습 CNN 전략의 한계를 극복하고 SOTA 성능을 달성했다. 더 나아가, PlantCLEF2022와 같은 대규모 도메인 특화 데이터셋이 식물 질병 진단과 같은 관련 하위 연구를 위한 강력한 사전 학습 리소스로 활용될 수 있음을 실험적으로 보여주었다.