Machine Learning Based Virtual Screening for Biodegradable Polyesters
논문 정보
- 제목: Machine Learning Based Virtual Screening for Biodegradable Polyesters
- 저자: Navya Nori (Milton High School)
- 학회/저널: Journal of Materials Science and Chemical Engineering
- 발행일: 2024-08-22
- DOI: 10.4236/msce.2024.128001
- 주요 연구 내용: 생성 모델(JTVAE)로 만든 수많은 폴리에스터 후보 분자들을 대상으로, 600개의 분자 실험 데이터로 학습된 Gradient Boosted Machine 모델을 사용하여 생분해성 점수를 예측함. 점수가 높은 분자들은 SynNet 모델을 통해 화학적 합성 가능성을 검증하여 최종 후보군을 선별하는 하이브리드 가상 스크리닝 프레임워크를 제안함.
- 주요 결과 및 결론: 개발된 생분해성 예측 모델은 테스트 데이터셋에서 84%의 AUROC와 87%의 AUPRC를 달성하여 높은 정확도를 보였음. 분자 구조 분석 결과, 짧은 탄소 골격, 에스터 결합, 적절한 소수성이 생분해성에 긍정적인 영향을 미치는 것으로 나타났으며, 최종 후보 분자들은 실험실에서 합성이 가능한 것으로 확인됨.
- 기여점: 기존의 고비용, 저효율 스크리닝 방법을 대체할 수 있는 저렴하고 확장 가능한 기계 학습 기반 접근법을 제시함. 생화학적 복잡성을 효과적으로 포착하여 새로운 생분해성 폴리에스터 후보 물질을 신속하게 발굴하고, 그 설계 원리에 대한 해석 가능성을 높임.
요약
초록
기존 폴리에스터는 분해에 200년 이상 소요되어 지속 가능한 대체재 개발이 시급하다. 이 연구는 최근 실험 데이터를 기반으로 학습된 모델이 분자의 생분해 가능성을 정확하게 예측할 수 있는지 평가한다. 600개 분자 데이터셋으로 학습된 Gradient Boosted Machine 분류 모델은 생분해성 예측에서 84%의 AUROC와 87%의 AUPRC를 달성했다. 또한 분자 구조, 결합 유형, 용해도와 같은 화학적 특성이 생분해성에 미치는 영향을 분석하고, 합성 트리 생성 모델(SynNet)을 통해 최종 후보 분자의 화학적 합성 가능성을 검증했다. 이 예측 및 화학적 규칙 해석을 통한 필터링 접근법은 저렴하고 확장성이 높으며, 섬유 산업 등에서 발생하는 환경 스트레스를 줄일 수 있는 새로운 폴리에스터 후보를 발굴하는 데 기여할 수 있다.
서론
의류 산업의 성장과 패스트 패션의 유행으로 폴리에스터 사용량이 급증하면서 막대한 양의 섬유 폐기물이 발생하고 있다. 특히 가장 많이 사용되는 PET는 자연 분해에 매우 오랜 시간이 걸려 환경 문제를 심화시킨다. 기존의 분자 구조 생성 기술(예: JTVAE)은 수많은 신규 화합물을 만들 수 있지만, 이를 스크리닝하는 고속 처리 스크리닝(HTS)이나 지식 기반 필터링 방식은 비용이 높고, 확장성이 부족하거나, 지나치게 제한적인 규칙으로 인해 잠재력 있는 후보를 놓치는 한계가 있었다. 본 연구는 이러한 문제들을 해결하기 위해 기계 학습을 활용한 효율적인 가상 스크리닝 방법을 제안한다.
모델 아키텍처 / 방법론
-
핵심 구조/방법: 연구는 3단계로 구성된다.
- 폴리머 생성: JTVAE 모델을 사용해 10,000개의 폴리머 분자를 생성하고, 이 중 에스터 결합을 가진 폴리에스터를 선별한다.
- 생분해성 예측: 600개의 기존 폴리머 생분해성 실험 데이터를 기반으로 Gradient Boosted Tree 모델을 학습시킨다. 이 모델은 분자 구조를 128비트 Morgan 핑거프린트로 변환하여 입력받고, 생성된 폴리에스터들의 생분해성 점수를 계산한다.
- 분석 및 검증: 점수가 가장 높은 상위 10개 폴리에스터에 대해 화학적 특성(분자 구조, 결합, 소수성)과 생분해성의 상관관계를 분석한다. 마지막으로 SynNet 모델을 이용해 이 분자들이 실제 합성이 가능한지 검증한다.
-
주요 구성 요소:
- 분자 생성: Junction Tree Variational Auto-Encoder (JTVAE)
- 생분해성 예측 모델: Gradient Boosted Machine
- 분자 표현: 128-bit Morgan Fingerprints
- 합성 가능성 검증: SynNet
-
수식: 본 논문에서는 구체적인 핵심 수식 대신 모델의 구조와 프로세스에 중점을 두어 설명한다.
-
알고리즘: 데이터는 70% 학습, 10% 검증, 20% 테스트용으로 분할하여 모델을 학습하고 평가했다. 검증 손실이 10회 이상 개선되지 않으면 학습을 조기 종료하는 방식을 사용했다.
실험 결과
- 주요 데이터셋: 예측 모델은 Fransen 등이 실험적으로 생분해성을 측정한 600개의 폴리에스터 및 폴리카보네이트 데이터셋으로 학습되었다.
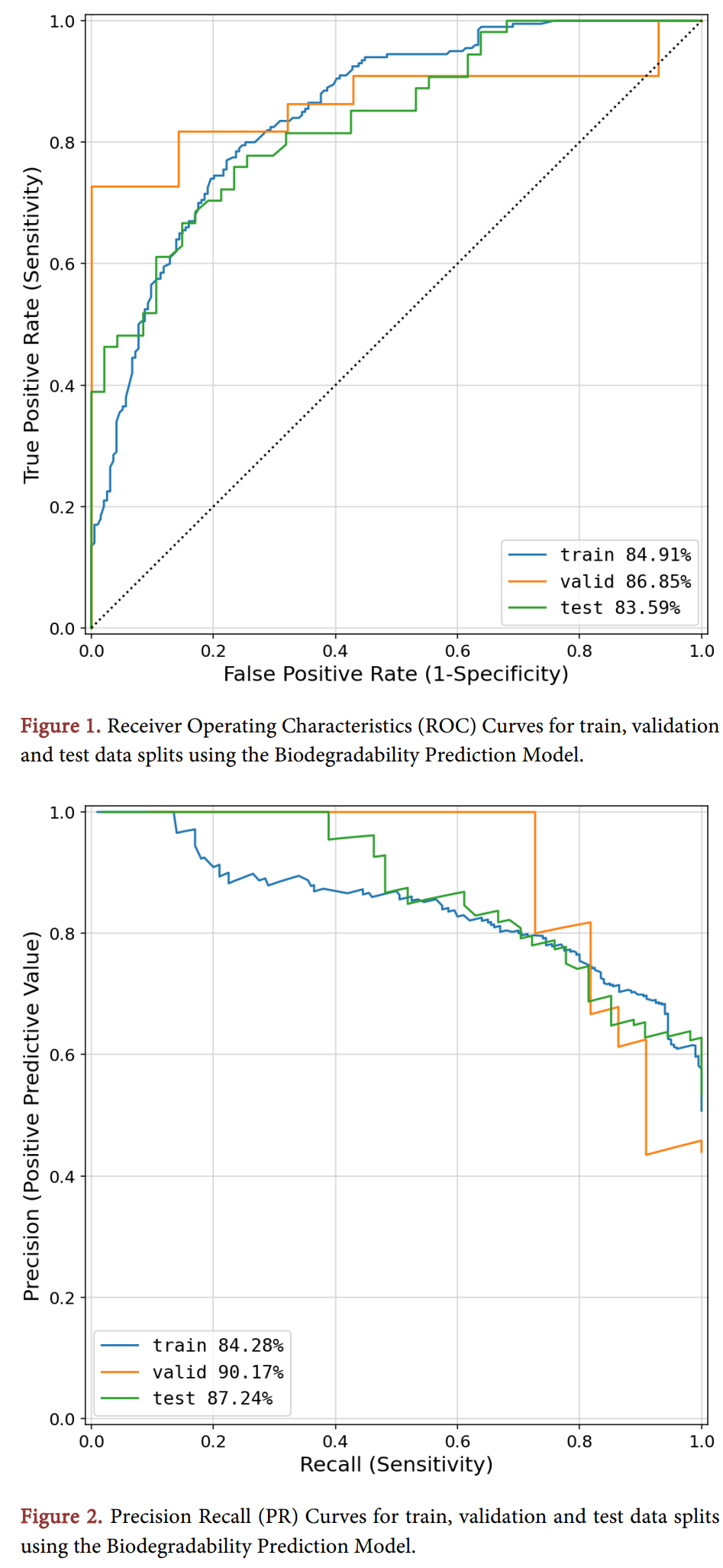
- 핵심 성능 지표: 생분해성 예측 모델은 테스트 데이터에서 83.59%의 AUROC와 87.24%의 AUPRC를 기록했다. 논문의 Figure 1(ROC 곡선)과 Figure 2(Precision-Recall 곡선)는 모델이 생분해 및 비생분해 분자를 효과적으로 구별함을 시각적으로 보여준다.
- 비교 결과:
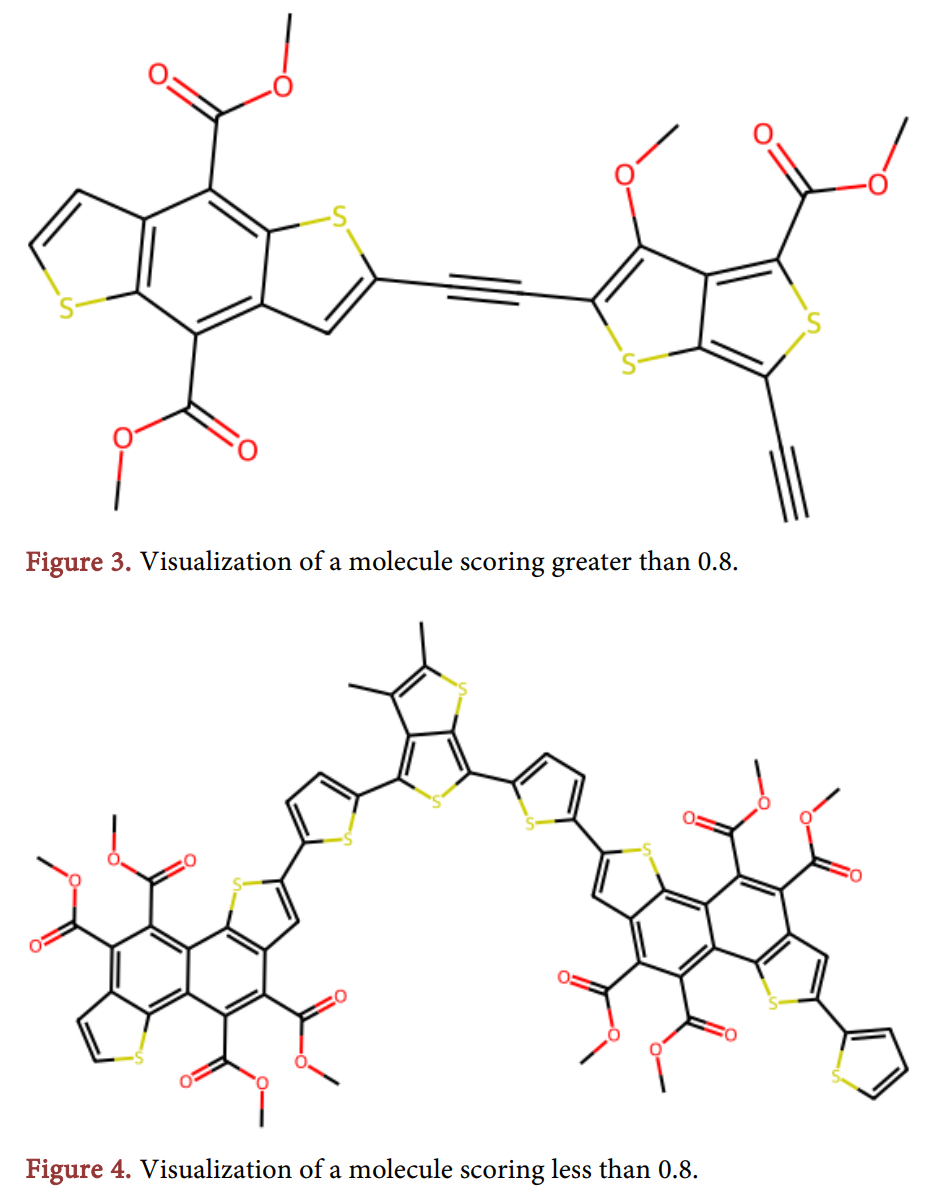
- 분자량: 탄소 골격이 짧을수록(7개 이하) 생분해성이 높았고, 분자량이 클수록 생분해성은 저해되었다. 논문의 Figure 3과 Figure 4는 각각 높은 점수와 낮은 점수를 받은 분자의 구조를 시각적으로 비교하여 이를 뒷받침한다.
- 결합 유형: 에스터 결합(fr_ester)은 생분해성과 0.062의 양의 상관관계를 보였다. 이는 에스터 결합의 극성으로 인해 가수분해 효소에 의해 쉽게 절단되기 때문이다.
- 소수성 (LogP): LogP로 측정된 소수성은 생분해성과 0.16의 약한 양의 상관관계를 보였다. 이는 소수성 분자가 미생물의 세포막을 더 쉽게 통과하여 분해될 가능성을 시사한다.
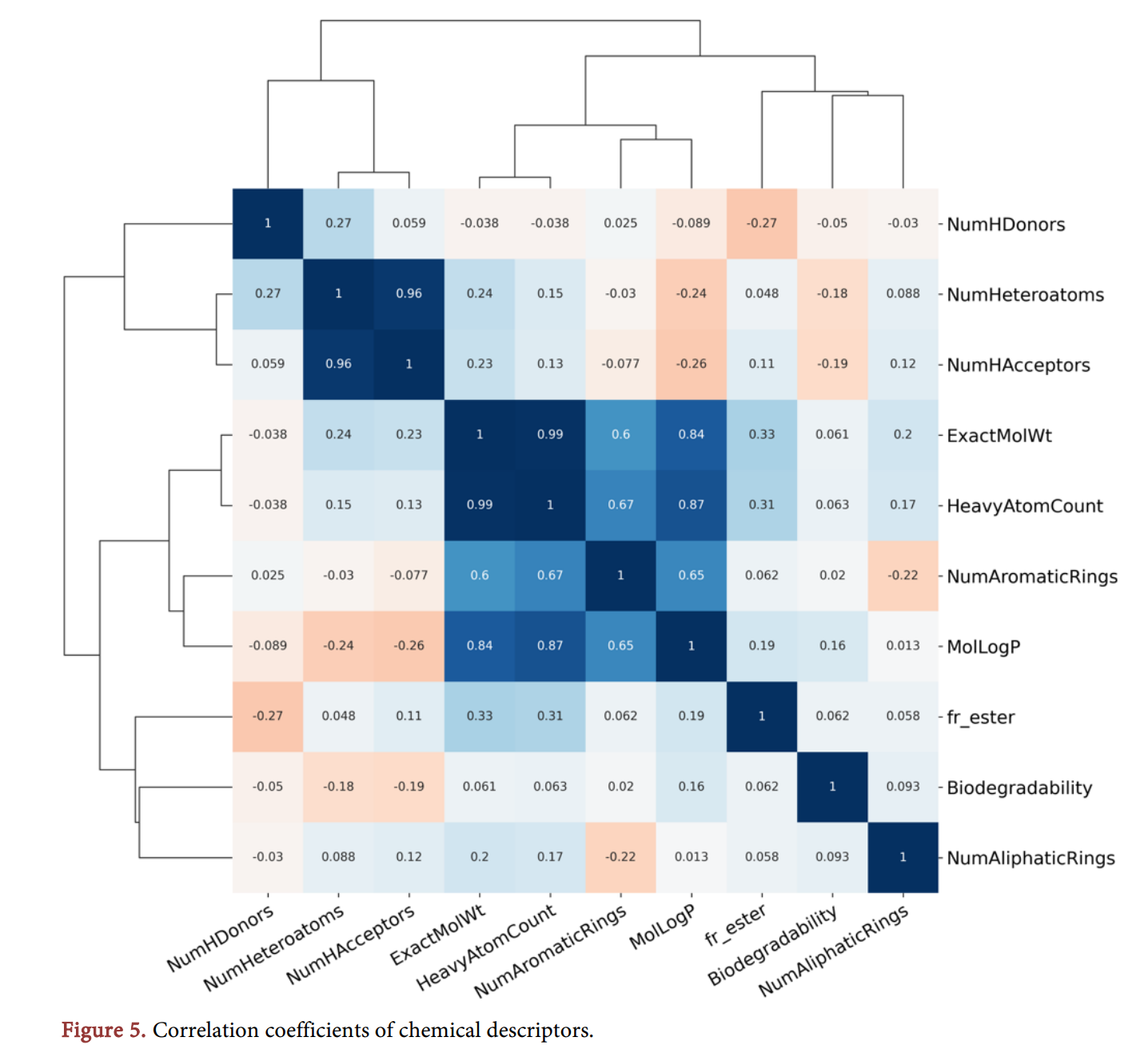
- 논문의 Figure 5는 이러한 화학적 특성과 생분해성 간의 상관관계 행렬을 보여준다.
- 합성 가능성: SynNet 분석 결과, 높은 점수를 받은 분자들은 모두 단순한 구조의 단량체들로부터 합성이 가능한 것으로 확인되었다.
결론
이 연구는 기계 학습을 이용해 생분해성 폴리에스터를 효과적으로 스크리닝하는 새롭고 실용적인 방법을 성공적으로 제시했다. 제안된 모델은 높은 정확도로 생분해성을 예측했으며, 분자 구조, 결합 특성, 소수성 등이 생분해에 미치는 영향을 분석하여 결과의 해석 가능성을 높였다. 또한, 최종 후보 분자들은 합성이 가능함이 검증되어 실제 산업 적용 가능성을 보였다. 향후 연구로는 용해도 등 다른 지속 가능성 관련 특성을 예측하는 모델을 추가로 개발하고, 가상 시뮬레이션 및 실제 실험(in vitro)을 통해 검증을 심화할 필요가 있다.