MUST-CNN: A Multilayer Shift-and-Stitch Deep Convolutional Architecture for Sequence-Based Protein Structure Prediction
논문 정보
- 제목: MUST-CNN: A Multilayer Shift-and-Stitch Deep Convolutional Architecture for Sequence-Based Protein Structure Prediction
- 저자: Zeming Lin, Jack Lanchantin, Yanjun Qi (University of Virginia)
- 학회/저널: Thirtieth AAAI Conference on Artificial Intelligence (AAAI-16)
- 발행일: 2016-02-12
- DOI: 해당 없음
- 주요 연구 내용: 단백질의 아미노산 서열로부터 2차 구조나 용매 접근성 같은 속성을 예측하기 위해 딥 컨볼루션 신경망(CNN)을 활용. Max-pooling으로 인한 해상도 저하 문제를 해결하기 위해, 다중 계층에 'shift-and-stitch' 기법을 적용하여 전체 서열에 대한 완전 밀집(fully dense) 예측을 효율적으로 생성하는 종단간(end-to-end) 모델 MUST-CNN을 제안함.
- 주요 결과 및 결론: 제안된 MUST-CNN 모델은 기존의 최첨단 모델들보다 구조적으로 더 단순함에도 불구하고, 4prot 및 CullPDB라는 두 개의 대규모 단백질 속성 예측 데이터셋에서 더 우수한 성능을 달성함. 특히 4prot 데이터셋에서 3클래스 2차 구조 예측(ssp) 정확도() 89.6%를 기록했으며, CullPDB 데이터셋에서는 8클래스 예측() 정확도 68.4%를 달성하여 기존 최고 성능을 경신함.
- 기여점: 딥 CNN에 적용 가능한 새로운 'multilayer shift-and-stitch' (MUST) 기법을 제안하여 학습 및 추론 시간을 크게 단축시키고 모델의 규모를 확장함. 또한, 임의 길이의 서열에 대해 각 위치별(per-position) 레이블링을 수행하는 일반적인 종단간 시스템을 제안했으며, 이를 통해 두 개의 대규모 단백질 데이터셋에서 최첨단 성능을 달성함.
요약
초록
단백질의 1차 아미노산 서열로부터 용매 접근성이나 2차 구조와 같은 속성을 예측하는 것은 생물정보학에서 중요한 과제이다. 최근 일부 딥러닝 모델이 전통적인 윈도우 기반 다층 퍼셉트론(MLP)을 능가하고 있다. 본 연구는 이미지 분류 분야에서 영감을 받아 단백질 속성 예측을 위한 딥 컨볼루션 신경망 아키텍처인 MUST-CNN을 제안한다. 이 아키텍처는 새로운 'multilayer shift-and-stitch' (MUST) 기법을 사용하여 단백질 서열의 각 위치에 대한 완전 밀집 예측을 생성한다. 제안 모델은 기존 최첨단 모델보다 훨씬 간단하지만 더 나은 결과를 달성하며, MUST 기법과 효율적인 컨볼루션 연산을 결합하여 훨씬 더 많은 파라미터를 사용하면서도 매우 빠른 예측 속도를 유지한다.
서론
단백질의 속성은 1차 아미노산 서열에 의해 거의 유일하게 결정된다고 알려져 있지만, 이를 대규모로 예측하는 것은 계산적으로 어렵다. 기존의 다층 퍼셉트론(MLP) 기반 방법들은 '윈도우(windowing)' 방식을 사용하는데, 이는 특정 아미노산과 그 주변 아미노산들만 입력으로 받아 하나의 레이블을 예측하는 과정을 반복한다. 이 방식은 학습 시간이 길고, 윈도우 크기가 작아 장거리 상호작용을 포착하기 어렵다는 단점이 있다. 이러한 문제를 해결하기 위해, 전체 서열을 한 번에 처리할 수 있는 컨볼루션 신경망(CNN) 기반의 접근법을 제안한다.
배경
기존의 단백질 속성 예측 알고리즘인 PSIPRED나 Jpred는 윈도우 방식을 사용하는 MLP 네트워크에 의존해왔다. CNN은 파라미터 공유와 풀링(pooling)을 통해 계산 효율성을 높이고 병렬화가 용이하여 MLP보다 훨씬 빠른 속도를 낼 수 있다. 하지만 CNN에서 풀링 연산은 출력의 해상도를 감소시켜 각 위치별로 레이블을 예측하는 것을 어렵게 만든다. 이 문제를 해결하기 위한 기법이 필요하다.
모델 아키텍처 / 방법론
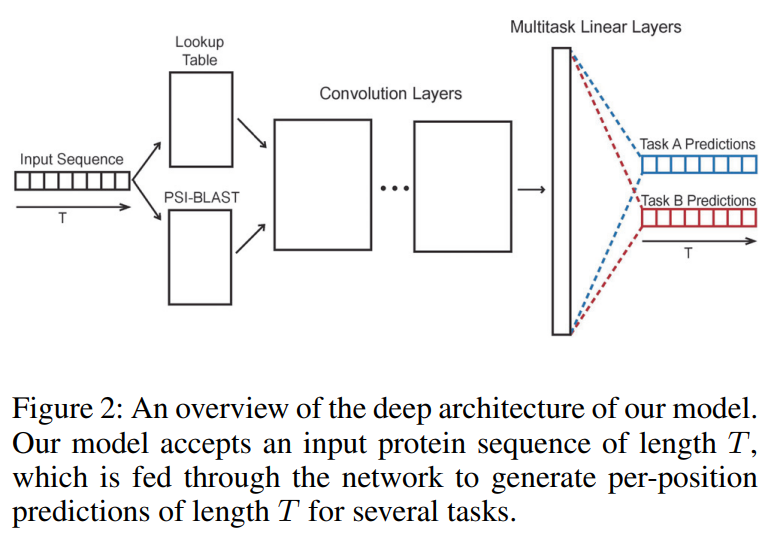
-
핵심 구조/방법: 논문에서 제안하는 MUST-CNN의 전체적인 구조는 논문의 Figure 2에 잘 나타나 있다. 모델은 아미노산 서열과 PSI-BLAST 프로파일을 입력으로 받아 임베딩을 위한 Lookup Table을 거친 후, 여러 개의 컨볼루션 계층을 통과한다. 최종적으로 다중 작업(multitask) 선형 계층을 통해 각 속성에 대한 예측을 동시에 출력한다. 이 구조의 핵심은 Multilayer Shift-and-Stitch (MUST) 기법이다.
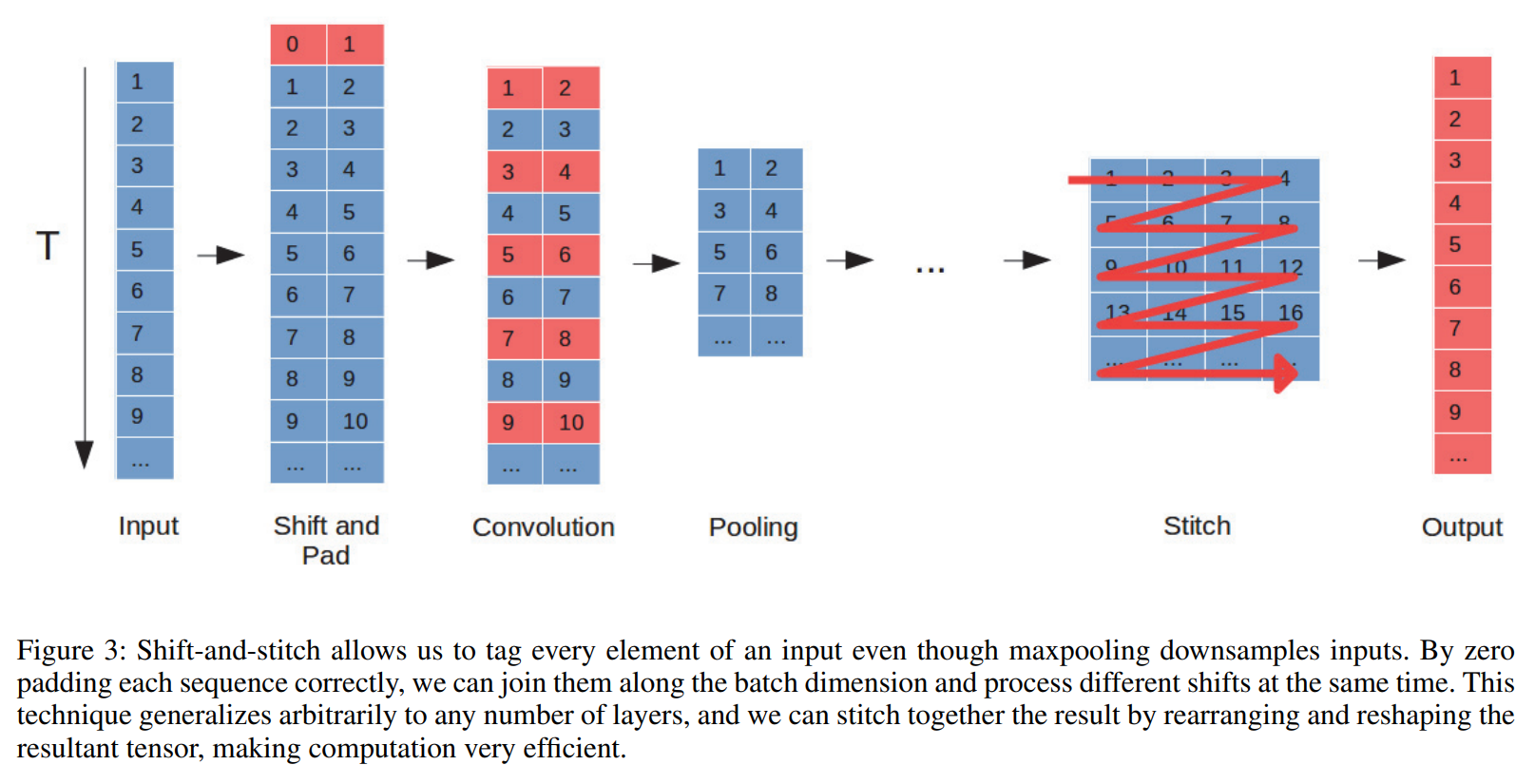
-
주요 구성 요소:
- CNN 계층: 1D 컨볼루션, 비선형 활성화 함수(ReLU), Max-pooling, 그리고 정규화를 위한 Dropout으로 구성된다.
- Multilayer Shift-and-Stitch (MUST): Max-pooling은 특징을 추출하고 번역 불변성(translation invariance)을 학습하는 데 중요하지만, 서열의 길이를 줄여 위치별 예측을 어렵게 한다. MUST는 이 문제를 해결하기 위해 고안되었다. 풀링 크기(m)만큼 입력 서열을 복제하고, 각 복제본을 미세하게 이동(shift)시킨 후 패딩(padding)한다. 이들을 하나의 배치로 묶어 CNN을 통과시킨 후, 결과로 나온 m개의 축소된 서열들을 다시 원래 순서대로 재조합(stitch)하여 완전한 해상도의 출력을 얻는다. 이 과정은 논문의 Figure 3에 시각적으로 설명되어 있다.
- 종단간 학습: 모델은 다중 작업 학습 프레임워크를 사용하여 여러 속성(2차 구조, 용매 접근성 등)을 동시에 예측하도록 학습된다.
-
수식: 모델은 모든 학습 데이터와 모든 작업에 대해 음의 로그 우도(negative log-likelihood) 함수를 최소화하도록 학습된다. 손실 함수는 다음과 같다. 여기서 는 시퀀스, 는 작업, 는 시퀀스 내 위치를 나타낸다.
-
알고리즘: 학습은 모멘텀을 이용한 확률적 경사 하강법(SGD)을 통해 이루어진다. 다중 작업 모델의 공동 학습이 끝난 후, 각 개별 작업에 대해 학습된 가중치를 초기값으로 사용하여 더 낮은 학습률로 미세 조정(fine-tuning)을 수행하여 성능을 극대화한다.
실험 결과
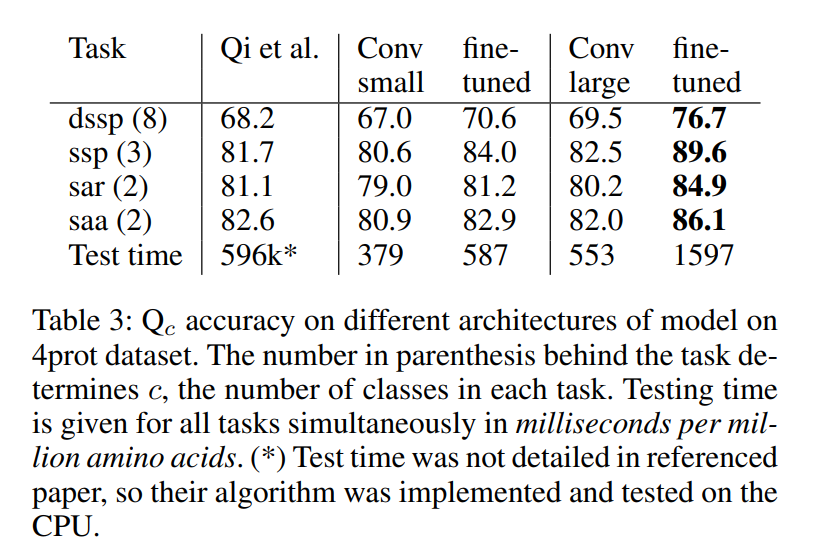

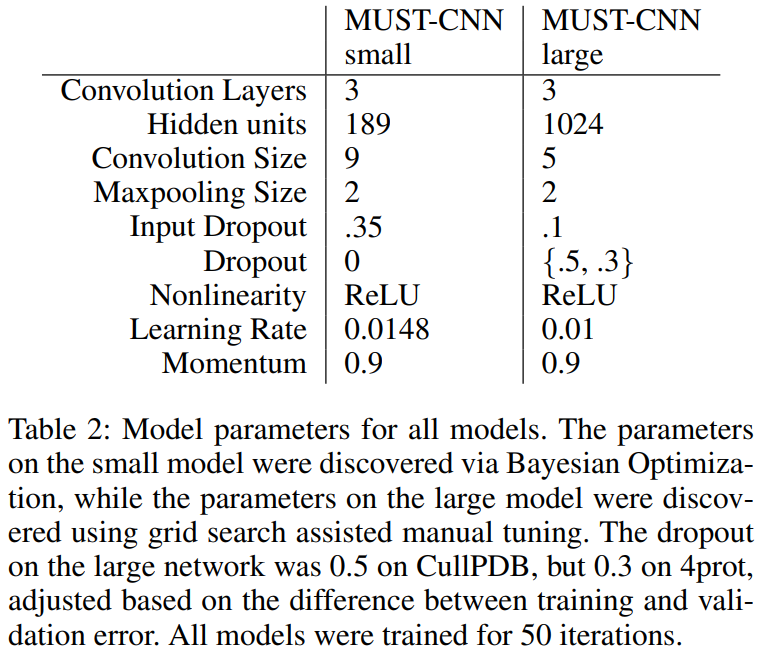
- 주요 데이터셋: 4prot와 CullPDB라는 두 개의 대규모 데이터셋을 사용했다. CullPDB 데이터셋으로 학습한 모델의 평가는 CB513 테스트셋을 사용했다.
- 핵심 성능 지표: 각 클래스별 위치 예측 정확도()를 사용한다. 3개 클래스 예측의 경우 , 8개 클래스 예측의 경우 로 표기한다.
- 비교 결과:
- 4prot 데이터셋: 미세 조정한 대형(large) MUST-CNN 모델은 3클래스 2차 구조 예측()에서 89.6%, 8클래스 예측()에서 76.7%의 정확도를 달성하여, 기존 SOTA였던 Qi et al. (2012)의 81.7%(), 68.2%()를 크게 상회했다 (논문의 Table 3).
- CullPDB/CB513 데이터셋: MUST-CNN은 정확도 68.4%를 기록하여, GSN (66.4%), LSTM (67.4%) 등 기존의 전체 서열 기반 모델들보다 더 나은 성능을 보였다 (논문의 Table 5).
- 속도: 제안된 모델은 백만 개의 아미노산을 2초 이내에 예측할 수 있을 정도로 매우 빠른 추론 속도를 보였다.
결론
본 연구는 단백질 서열 예측을 위해 'multilayer shift-and-stitch' 기법을 적용한 딥 컨볼루션 아키텍처인 MUST-CNN을 제안했다. 이 모델은 구조적으로는 더 복잡한 기존 모델들(GSN, LSTM 등)보다 간단하지만, 연산 속도의 이점을 활용해 더 큰 모델 용량을 확보함으로써 더 우수하거나 동등한 성능을 달성했다. 제안된 모델은 두 개의 다른 대규모 데이터셋에서 약간의 하이퍼파라미터 조정만으로도 견고하게 좋은 성능을 보여주었으며, 이 기법은 단백질 예측 외 다른 일반적인 서열 태깅 문제에도 적용될 수 있을 것이다.